【导读】2015年10月5日,瑞典斯德哥尔摩,诺贝尔委员会举办新闻发布会,宣布2015年诺贝尔生理学或医学奖得主。药学家屠呦呦,爱尔兰科学家威廉·坎贝尔、日本科学家大村智分享该奖项。

图1 2015诺贝尔生理学或医学奖得主
至此,屠呦呦成为位获得诺贝尔科学类奖项的科学家、位获得诺贝尔生理医学奖的华人科学家。屠呦呦发现的对抗疟疾的神药青蒿素也引起举世瞩目。
青蒿素的发现表明,中医药是一个伟大的宝库,有宝贵的财富,需要我们去发现、挖掘和研究。而屠呦呦此次因其发现青蒿素的突出贡献获得诺贝尔奖也为中药发展迎来新的契机。

图2 药学家屠呦呦肖像照和其工作描述图
创腾科技作为的生命科学信息提供商,基于数据库、分子模拟与分子设计平台Discovery Studio以及强大的信息整合和流程定制的科学平台Pipeline Pilot,能够为中药研发的科学化和现代化助上一臂之力。
中药化合物数据信息的提供
·中药化学数据库
· 致突变型(Mutagenicity(Ames test))
· 啮齿动物致癌性(Rodent Carcinogenicity)包括 NTP及FDA 数据集
· 大鼠长期口服zui低毒副反应水平(Rat Chronic Oral Lowest Observed Adverse Effect Level, LOAEL)
· 皮肤致敏性(Skin Sensitization
· 黑头呆鱼LC50(Fathead Minnow LC50)
· 大型溞EC50(Daphnia Magna EC50)
· VlogP
· 眼刺激性(Ocular Irritation)
· 大鼠吸入LC50(Inhalational LC50)
· 好养生物降解性能(Aerobic Biodegradability)

图6 青蒿素的ADMET Descriptors预测结果
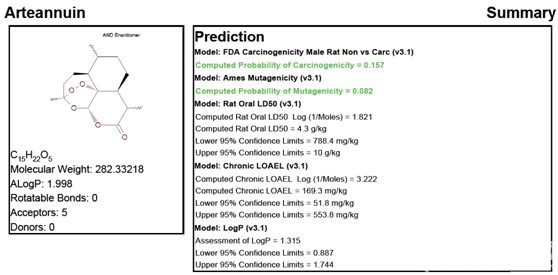
图7 青蒿素的TOPKAT预测结果
中药靶向原理、有效成分的预测
中药的现代化,应该是真正理解其有效成分、药效机理、靶向原理,知其然并知其所以然。
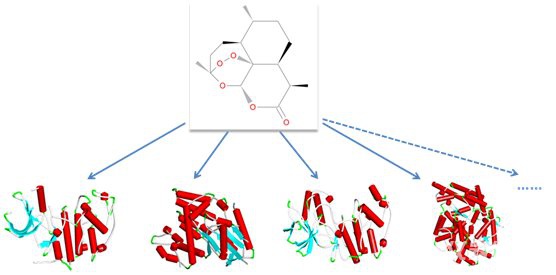
图8 青蒿素潜在靶标的预测示意图
对于未知靶标的中药有效成分,可以基于如下三种方法进行反向找靶,从而预测其作用机制。
·药效团模型搜索(Compound Profiling)
Discovery Studio为研究者提供基于受体或基于受体-配体相互作用构建代表受体活性口袋化学和几何信息的药效团模型的算法。
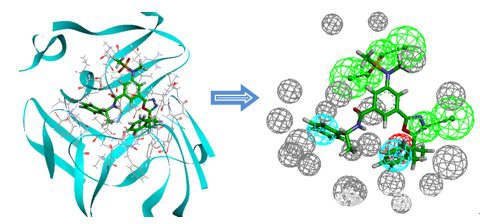
图9基于受体-配体复合物产生药效团
基于药效团模型来搜索潜在靶标的方法,就是将中药有效成分与多个代表各靶标蛋白的药效团模型相互匹配,终按照匹配打分的高低来判定潜在作用靶标。Discovery Studio中包含目前市场上zui大的受体-配体复合物药效团数据库PharmaDB,该数据库是基于scPDB(2012)中7028个复合物晶体结构构建的,共含117423个药效团模型,并且这些模型已根据不同的靶标类型进行了分类。Discovery Studio中自带的流程Ligand Profiler可自动实现多个分子和多个药效团模型的快速匹配并进行匹配度打分排序。因此,结合PharmaDB,Discovery Studio可以快速有效且地进行靶标搜寻、中草药有效成分的确定以及毒副作用评价。
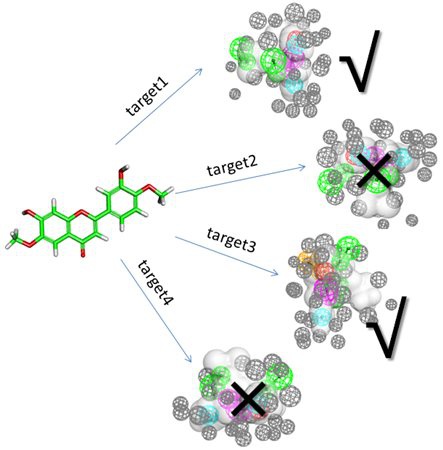
图10基于药效团模型的反向找靶示意图,“对号”代表命中的模型
·反向分子对接(Target Fishing)
传统的分子对接方法可以帮助科研工作者预测靶标分子与待研化合物的相互作用模式,并借助打分函数评价分子的构效关系。然而,借助计算流程编辑与管理平台Pipeline Pilot以及分子模拟平台Discovery Studio中的分子对接算法、打分函数,创腾科技为医药研究者提供基于分子对接方法的化合物反向找靶策略。

图11基于分子对接的化合物反向找靶计算流程图
整个设计思路分为三个步骤:
1. 读取用户的小分子结构
· 读入的文件格式可能不同:sdf、mol、mol2、skc等
· 实现读入小分子的二维/三维结构转化,结构标准化,加氢,结构优化等
2. 反向对接及打分(等于多个正向对接)
· 遍历蛋白数据库文件,获得每个蛋白的文件路径和结合位点
· LibDock参数自动设置和填写(图示流程整合了DS中的LibDock对接模块,如需要,也可替换其它对接程序)
· 自动循环,使小分子与每个蛋白受体对接
· 打分、筛选和排序
3 结果报表输出
· 柱状图显示靶标打分和终排序
· 对接结构和打分情况
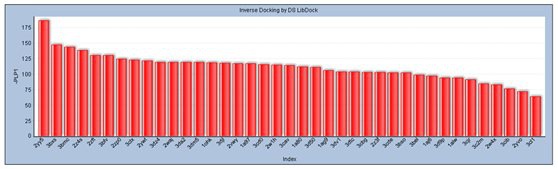

图12基于DS+PP反向找靶流程的结果示意
其中,靶标可来源于scPDB数据库(http://bioinfo-pharma.u-strasbg.fr/scPDB ),该数据库收集了标准PDB数据库中含有药物结合位点的蛋白,可根据配体、蛋白、结合方式为特征进行搜索。
·基于小分子相似性分析
Discovery Studio为研究者提供基于分子指纹的分子结构相似性搜索,即DS可以计算中药活性成分与已包含化合物生物活性以及靶标注释的化合物数据库中小分子化合物的Tanimoto系数等,从而进行相似性评价,进而预测其潜在靶标。如果输入的分子能够在数据库中搜索到它本身,则可以获得其已知靶标;如果输入的分子能够在数据库中搜索到与其相似的化合物,则根据与其结构相似的分子靶标可推测输入分子的靶标信息。
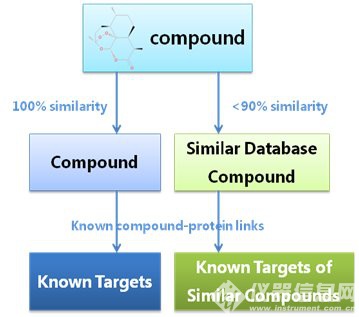
图13 根据同已有靶标分子相似性的分析进行未知分子靶标的预测流程示意图